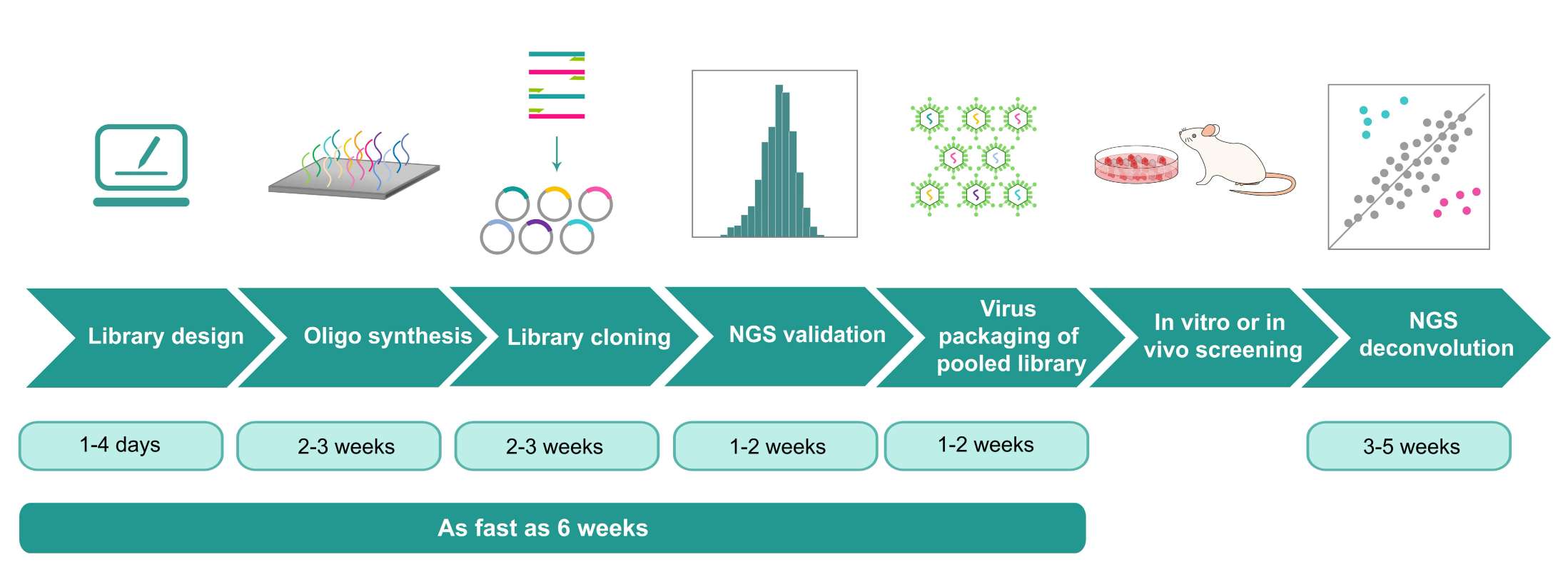
技术详情
突变文库的构建策略
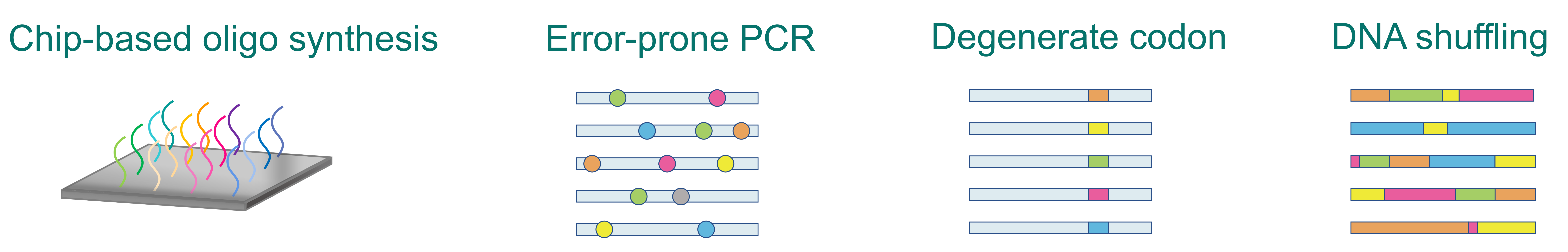
芯片合成寡核苷酸
芯片合成寡核苷酸的方式非常适合用于生成的具有各种大小和序列的突变体,比如可在给定位点制造饱和突变。我们基于芯片的 DNA 合成长度通常可达300 nt,且错误率较低。
易错PCR
易错PCR是最直接用于开发高度多样化的突变文库的选择,这其中包括从修改标准PCR方法到突变AAV衣壳基因等多种方式。更具体而言,易错PCR结合了多种次优化的PCR条件,如使用低保真性聚合酶、更长的延伸时间、更高的Mg2+浓度、添加Mn2+,以及使用不相等的多种dNTP浓度等方式来对AAV衣壳基因引入随机突变。
简并密码子
密码子简并机制允许一个氨基酸由多个密码子编码。通过向由寡核苷酸合成中的简并密码子引入了受控的、高度随机的突变,可对可能产生阳性突变的区域和氨基酸进行针对性的靶向。例如,广泛使用的简并密码子突变文库——NNK文库,其NNK简并引物涵盖了32种密码子组合(N= A/C/G/T,K= G/T),可以编码所有20种氨基酸和一个终止密码子。在文库构建中利用简并密码子是一种引入多样性的有效方法。
| 简并密码子类型 | 密码子组合数量 | 终止密码子数量 | 氨基酸 |
|---|
| NNN | 64 | 3 | All 20 |
| NNK / NNS | 32 | 1 | All 20 |
| NDT | 12 | 0 | RNDCGHILFSYV |
| DBK | 18 | 0 | ARCGILMFSTWV |
| NRT | 8 | 0 | RNDCGHSY |
DNA洗牌
DNA洗牌是一种通过同源重组产生嵌合DNA序列的高效方法。这项技术首先将亲本基因切割成随机片段,然后使用无引物的PCR技术并根据部分序列同源性进行重组,从而将它们重新组装成新的嵌合序列。另外,合成洗牌将合理设计与定向进化相结合,可用于创建高复杂性文库。在采用其他突变方法(如易错PCR)后也可以进行家族DNA洗牌。
实验数据
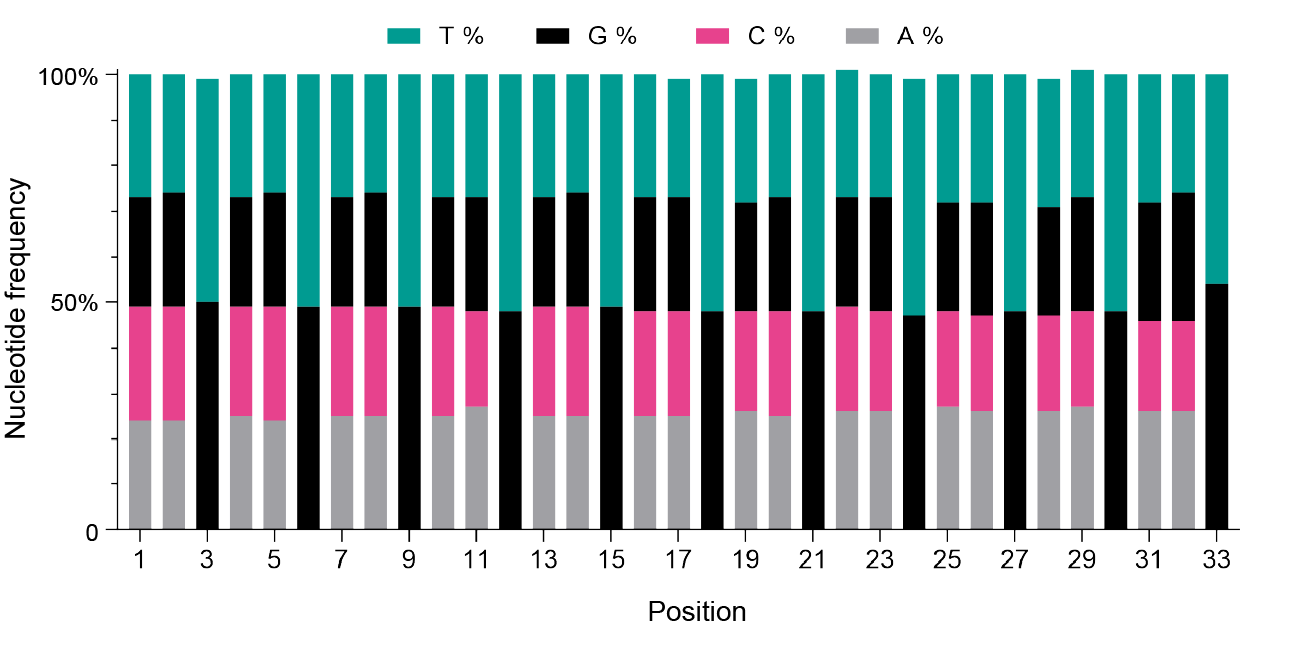
图1 质粒文库(NNK)11的核苷酸构成
该文库采用NNK简并密码子策略构建。每根柱子代表DNA序列中不同的核苷酸位置,颜色表示观察到的核苷酸类型。所有位置都显示出预期的核苷酸,且观察到的频率接近理论值(N = 25% A + 25% T +25% G + 25% C,K = 50% T + 50% G)。
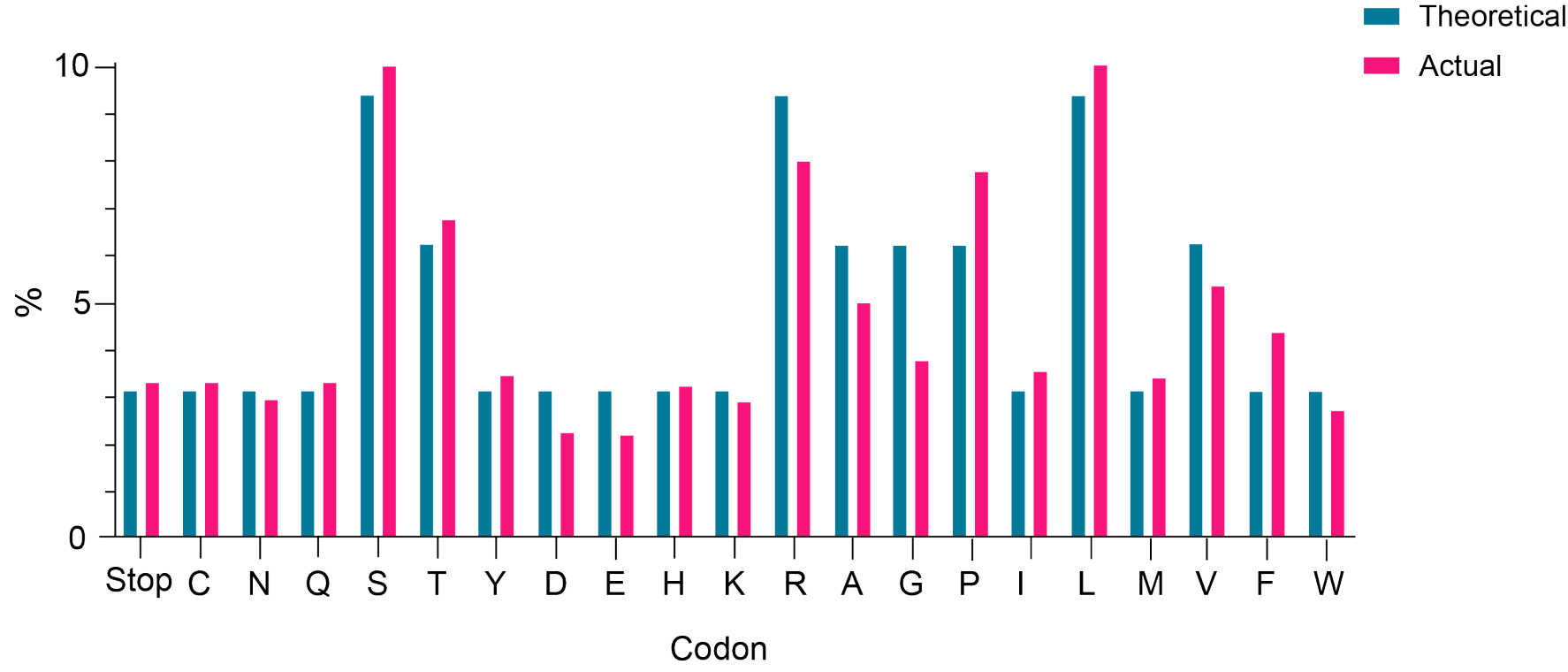
图2 质粒文库7-mer可变区的氨基酸分布
该文库利用NNK简并密码子策略构建而成。每根柱子分别代表理论(绿)/实际(红)上的特异氨基酸/终止密码子的百分比。观察到的20种氨基酸和终止密码子的实际频率与理论频率非常接近。
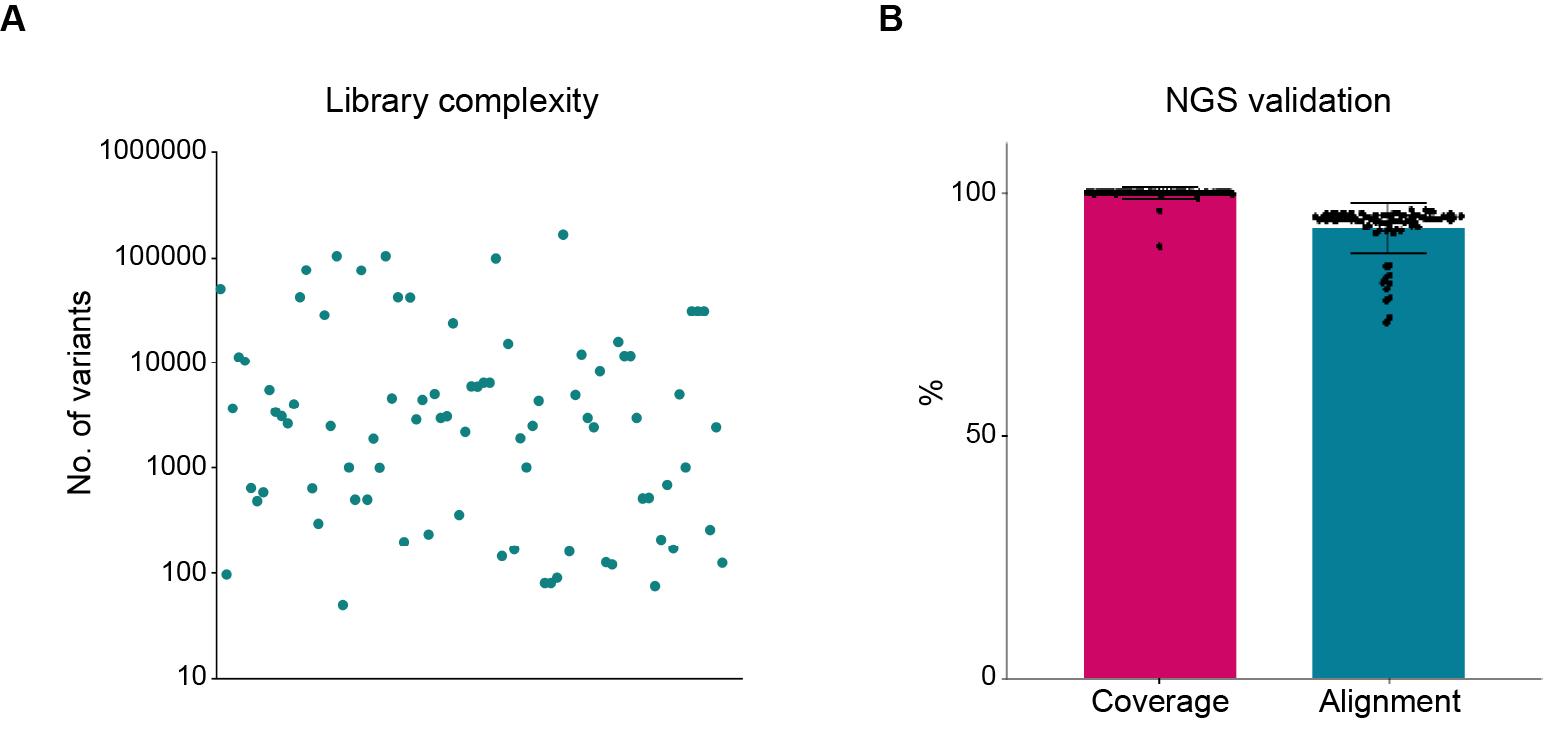
图3 云舟生物芯片合成的寡核苷酸文库。图示为复杂度、覆盖率和序列比对的一致性。
数据以无偏向的方式收集于云舟生物使用芯片合成的寡核苷酸文库。(A)这些定制文库的大小分布高度多样化,复杂度从102到超过105不等。(B)覆盖率和读取的比对率均很高接近100%,突出表明了我们文库的极高准确性和低错误率。